Kapitel 4 Wichtiges Grundlagenwissen
Bevor wir richtig loslegen können, müssen wir einige Grundlagen besprechen. Das Abspeichern von Zwischenergebnissen in Form von Variablen ist genauso essentiell wie die verschiedenen Datentypen, die in den jeweiligen Spalten deines Datensatzes enthalten sind. Auch einzelne Spalten mithilfe des Dollar-Operators aus einem Datensatz herauszuziehen ist ein zentrales Konzept, auf das wir im Verlauf dieses Buches häufig zurückgreifen werden.
4.1 Schritt für Schritt beginnen
1. Schritt: RStudio starten. Nach der Installation von R und RStudio (siehe Kapitel 2.1.1 und 2.1.2) kann RStudio in Windows durch Tippen der Windows-Taste mit anschließender Eingabe von RStudio und in macOS über den Finder gestartet werden. In der Konsole wird dabei automatisch die installierte R Version angezeigt.
Abbildung 4.1: Starten von RStudio.
2. Schritt: R Projekt erstellen. Als nächster Schritt sollte ein in sich abgeschlossenes Projekt erstellt werden (siehe Kapitel 3). Das erfolgreiche Erstellen und Öffnen des Projektes erkennt man daran, dass oben rechts in RStudio anstelle von Project: (None) der Name des Projektes steht (z.B. Beispiel). Manchmal wird, wie in Abbildung 4.2 gezeigt, der Überordner der Projektdatei mit angezeigt (hier der Ordner R Skripte). Wir sehen auch, dass unten rechts im Reiter Files jetzt die im Projektordner befindlichen Dateien angezeigt werden.
Abbildung 4.2: Innerhalb von RStudio ein neues R Projekt erstellen.
3. Schritt: R Skript erstellen. Da die Funktionsaufrufe in der Konsole nur einmalig ausgeführt und nicht gespeichert werden, muss anschließend das R Skript entweder durch Klicken des Pluszeichens links oben oder durch die Tastenkombination Strg / Cmd + Shift + N erstellt werden (siehe Kapitel 2.1.2).
Abbildung 4.3: Ein neues R Skript für die Analysen erstellen.
4. Schritt: Benötigte Packages laden. Bei jedem Start von RStudio müssen zunächst alle verwendeten Packages neu geladen werden. Dafür markieren wir die verschiedenen library() Aufrufe (hier vier Stück) und führen diese mit der Tastenkombination Strg / Cmd + Enter aus. Anders als in der Konsole werden die Funktion in einem R Skript also nicht alleine mit Enter sondern mit der Kombination Strg / Cmd + Enter ausgeführt. Um einzelne Zeilen auszuführen, genügt es, den Cursor auf der Zeile zu haben und dabei Strg / Cmd + Enter auszuführen. Manche Packages geben beim Laden Benachrichtigungen zurück. Was die Benachrichtigungen des tidyverse in Abbildung 4.4 bedeuten, wird in Kapitel 6 erläutert.
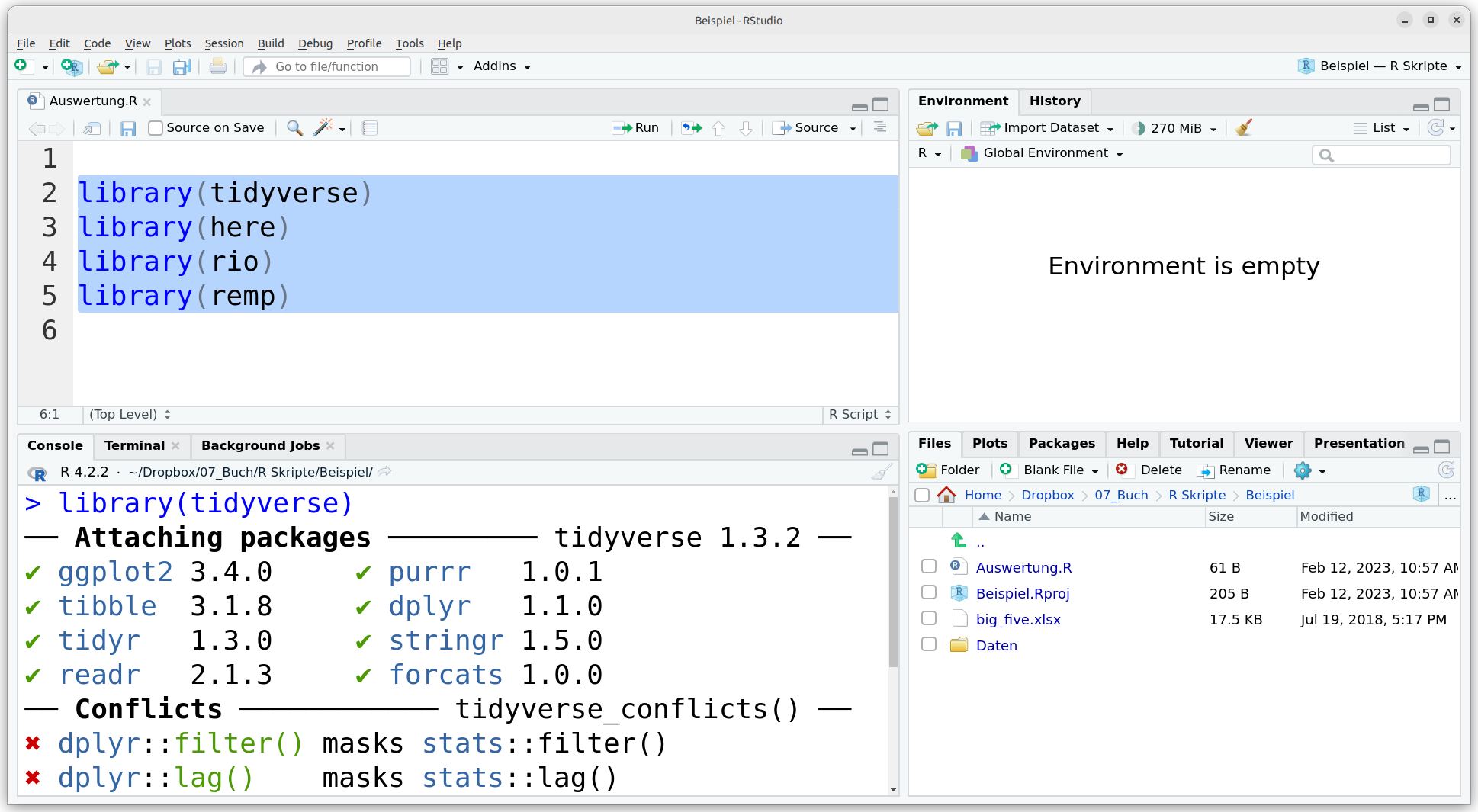
Abbildung 4.4: Packages nach jedem Start neu markieren und ausführen.
5. Schritt: Datensatz einlesen und Analysen durchführen. Nach Laden der Packages kann der Datensatz eingelesen werden (siehe Kapitel 5.1). Durch das Einlesen und Speichern mit Zuweisungspfeil erscheint der Datensatz rechts oben in der Environment (siehe Kapitel 4.2). Man kann also im weiteren Verlauf auf den big5 Datensatz namens daten zugreifen. Auch dieser muss bei Neustart von RStudio erneut durch Auswahl der Zeile (hier Zeile 7) und Betätigen der Tastenkombination Strg / Cmd + Enter eingelesen werden. Anschließend sollte man im Rahmen der weiteren Auswertung der Daten immer Kommentare einbauen, die mit einer führenden Raute markiert werden (siehe Zeile 9 in Abbildung 4.5). Exemplarisch schauen wir uns hier die Spalten Extraversion und Neurotizismus als ersten Schritt unserer Datenanalyse an (siehe Kapitel 6.2).
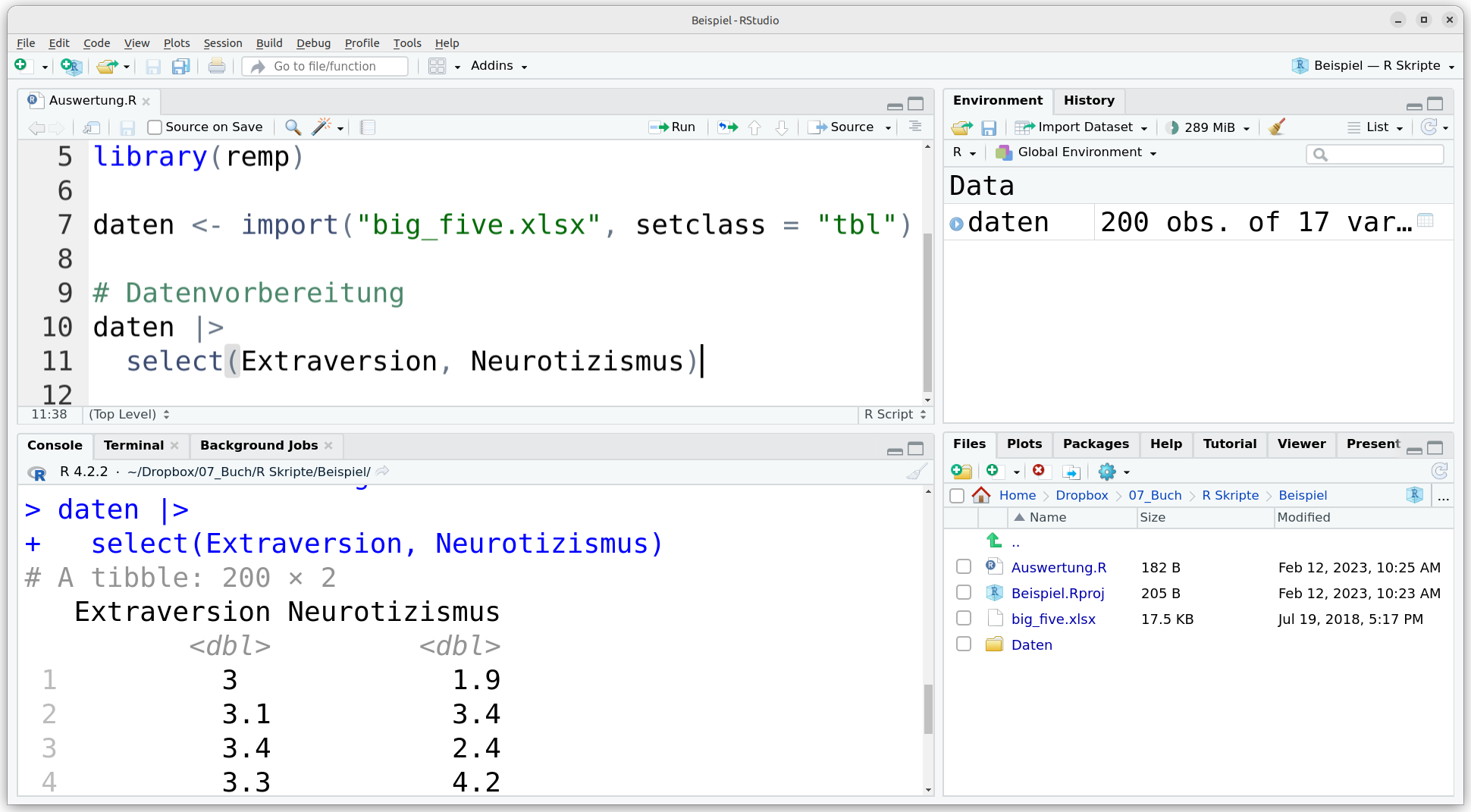
Abbildung 4.5: Datensatz einlesen und Analysen durchführen.
4.2 Variablen speichern und verwenden
Ein zentrales Konzept in R ist das Speichern von Variablen mithilfe des Zuweisungspfeils. Dies ist vor allem für all jene ungewöhnlich, die das Erstellen von Variablen aus anderen Programmiersprachen mit dem Gleichheitszeichen kennengelernt haben. Wenn man das Ergebnis der durchgeführten Operation nicht speichert, ist es sofort weg und muss erneut ausgeführt werden. Würde man nun
[1] 4rechnen, gibt R zwar 4 zurück, allerdings kann man später nicht mehr auf diese 4 zurückgreifen. Wenn man beispielsweise einen Datensatz einliest, ohne diesen mit dem Zuweisungspfeil zu speichern, kann man auf diesen im weiteren Verlauf nicht zugreifen. Mithilfe des Zuweisungspfeils wird die Variable in die lokale Environment gespeichert. Wir erinnern uns, die Environment ist in der Standardeinstellung nach Installation von RStudio im Fenster oben rechts. Möchten wir beispielsweise die Rechenoperation von vorhin namens rechnung speichern, würde man wie folgt vorgehen:
Im Nachfolgenden könnte man nun diese Variable jederzeit wieder aufrufen.
[1] 4Auch ist es nun möglich, weitere Rechenoperationen mit dem vorherigen Ergebnis auszuführen. Zum Beispiel könnte man unser Ergebnis namens rechnung mit der Zahl 4 multiplizieren.
[1] 16Variablen kann man grundsätzlich fast so benennen, wie man möchte. Man darf nur nicht mit einer Zahl anfangen oder nach einem Punkt direkt eine Zahl als Namen wählen wie bspw. bei .2VariablenName. Auf Umlaute sollte im Zusammenhang mit Programmiersprachen aufgrund verschiedener Zeichenkodierungen ebenfalls immer verzichtet werden. Groß- und Kleinschreibung macht ebenfalls einen Unterschied.
Achte beim Umgang mit Variablen immer auf die exakte Schreibweise beim Speichern in der Environment. Das Ergebnis als rechnung zu speichern ist nicht dasselbe wie als Rechnung oder rechnunG.
In den Variablen können sämtliche Datenstrukturen (siehe Kapitel 11) verstaut werden. Für den Moment reicht es für uns zu wissen, dass wir Datensätze und Zwischenergebnisse in den Variablen abspeichern müssen, um weiter darauf zugreifen zu können. Variablen können einfach überschrieben werden, indem man der Variable einen anderen Wert zuweist. Gerade in der Datenvorbereitung kann es schon einmal verlockend sein, die Änderungen unter demselben Variablennamen zu speichern. Wenn man allerdings eine unbeabsichtigte Änderung abspeichert, kann dies nicht rückgängig gemacht werden. Der Datensatz muss als Resultat erneut eingelesen werden. Es sei also vor allem am Anfang Vorsicht geboten.
Auf der anderen Seite sollte man auch nicht jeden einzelnen Schritt in der Datenvorbereitung mit einem bedeutungslosen Namen versehen. Schließlich hätten die Zwischenergebnisse rechnung1, rechnung2, rechnung3 und rechnung4 keine Information im Namen, welches Ergebnis nun welche Änderung enthält. Aussagekräftige Namen helfen anderen, deinen Code zu verstehen. Wenn du dir nun denkst, ohnehin nicht mit anderen zusammenarbeiten zu werden, lass dir gesagt sein: der oder die Andere bist in den meisten Fällen du selbst einige Wochen oder Monate später. Ohne Dokumentation und vernünftige Namensgebung sieht dein R Skript Monate später schnell so aus, als hätte es irgendein Fremder geschrieben. Dein zukünftiges Ich wird dir dankbar sein.
Behalte immer im Hinterkopf, dass der Zuweisungspfeil zwar die Variable speichert, aber keinen direkten Output in der Konsole ausgibt. Oft erweckt das den Eindruck, als wäre nichts passiert. Es hilft dann, den Namen der Variable, wie zuvor gezeigt, explizit aufzurufen.
Wenn man etwas im Code kommentieren möchte, muss eine führende Raute hinzugefügt werden. Das eignet sich nicht nur für kurze Beschreibungen, sondern auch als Gliederung eines langen R Skriptes. Beispielsweise könnte man so den Abschnitt der Berechnung vom Erstellen von Visualisierungen optisch trennen. Die Anzahl der Rauten spielt dabei keine Rolle.
[1] 64.3 Datentypen
Grundsätzlich gibt es in R vier verschiedene Grunddatentypen: Integer, Double, Character und Logical (siehe Abbildung 4.6). Dabei lassen sich Integer und Double zum Datentyp Numeric zusammenfassen, da die Unterscheidung dieser beiden in R selten von Bedeutung ist. Schauen wir uns die Datentypen nun etwas genauer an. Numeric (kurz: <num>) beschreibt numerische Werte, also Zahlen. Integer (kurz: <int>) sind ganze Zahlen und Doubles (kurz: <dbl>) Dezimalzahlen. Beachte dabei, dass Dezimalzahlen in R mit Punkten und nicht mit Kommata dargestellt werden.
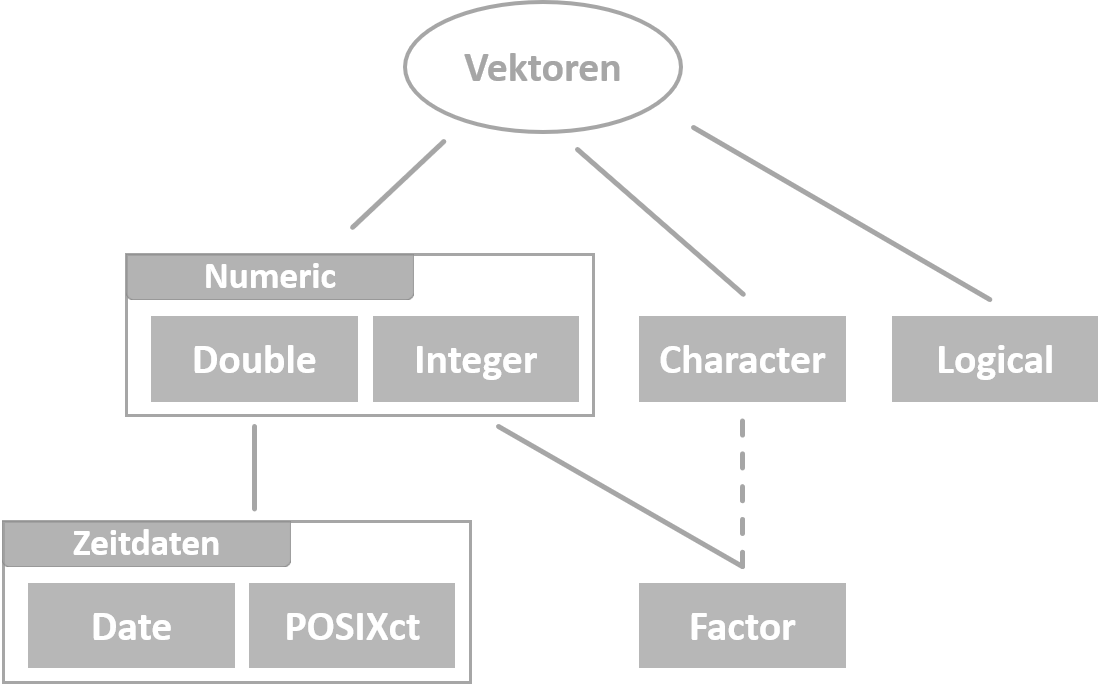
Abbildung 4.6: Schematische Übersicht über die wichtigsten Datentypen in R.
4.3.1 Zahlen, Buchstaben und logische Abfragen
Beispiele für Numerics wären zum Beispiel Alter, Gehalt oder der Blutdruck. Wenn wir später Datentypen aus einem echten Datensatz ansehen, wirst du schnell merken, dass beinahe alle Zahlen als Double deklariert werden. Das liegt an der Eigenheit von R, ein L hinter die Zahl setzen zu müssen, wenn es sich um eine ganze Zahl hat. Dies hat allerdings keinerlei Auswirkung auf die in diesem Buch vorgestellten Funktionen.
Double:
Integer:
Character (<chr>) ist der Datentyp, der Text enthalten kann – also einzelne Buchstaben, Zeichen, Wörter oder ganze Sätze. Dabei muss der Text immer in Anführungszeichen stehen. Solange die Anführungszeichen verwendet werden, kann mit Ausnahme vom Backslash (\) alles geschrieben werden. Beispiele für Characters wären zum Beispiel die Blutgruppe, das Herkunftsland oder Allergien.
Logical oder auch logische Datentypen sind etwas abstrakter und kommen in Datensätzen seltener vor. Sie werden dann benötigt, wenn wir aufgrund von bestimmten Bedingungen manche Operationen durchführen möchten und andere wiederum nicht. Beispiele dafür wären die Auswahl derjenigen Personen, die über 50 Jahre alt sind oder eine neue Spalte namens Geschlecht zu erstellen, die nur bei weiblichen Personen eine 1 und ansonsten eine 0 einträgt. Dabei wird nämlich geprüft, ob die Aussage (zum Beispiel Person A ist älter als 50) wahr oder falsch ist. Das heißt, es gibt dabei zwei Zustände: TRUE oder FALSE (immer in Großbuchstaben). Eine Bedingung kann entweder zutreffen oder eben nicht. Es gibt verschiedene Funktionen, die TRUE oder FALSE zurückgeben, auf die wir im Verlaufe des Buches noch stoßen werden. Grundsätzlich gibt es dabei nur wenige verschiedene Grundoperatoren, die man kennen sollte. Um zu schauen, ob zwei Werte gleich sind, benutzen wir ein doppeltes Gleichheitszeichen (==).
[1] TRUEGleiches Prinzip gilt für größer gleich (>=) und kleiner gleich (<=). Für größer (>) oder kleiner (<) reicht hingegen das einzelne mathematische Zeichen. Möchten wir nun logische Operationen kombinieren, verwenden wir UND (&) oder ODER (|). Bei UND müssen beide Aussagen wahr sein.
[1] FALSEIm Falle vom ODER-Operator muss nur eine logische Abfrage zutreffen, um ein TRUE ausgegeben zu bekommen. Man würde es wie folgt lesen: Entweder ist 1 kleiner 2 ODER 1 ist gleich 2. Da die erste Aussage wahr ist, wird TRUE zurückgegeben.
[1] TRUENeben den logischen Operatoren & und | existieren in R ebenfalls die doppelten Varianten && und ||. Der Unterschied besteht in dem Ausgabeformat. Im Falle der einzelnen Operatoren (&, |) können (vektorisiert) unbegrenzt viele Abfragen überprüft werden. Der Ausdruck c("A", "C") == "A" gibt bspw. TRUE FALSE zurück, da der erste Buchstabe ein A ist und der zweite nicht. Bei den doppelten Operatoren (&&, ||) würde dieser Ausdruck einen Fehler produzieren, da hier die logische Abfrage immer nur einen einzigen logischen Wert zurückgeben darf (entweder TRUE oder FALSE). Dies ist vor allem bei if-Abfragen oder while-Schleifen nützlich. Im Kontext der wissenschaftlichen Datenanalyse greifen wir in den meisten Fällen auf die einzelnen logischen Operatoren zurück (&, |).
So können beliebig viele logischen Operationen miteinander kombiniert werden. Ein besonderer Fall logischer Datentypen ist NA (Akronym für Not Available), also die Bezeichnung für einen fehlenden Wert. Wir können einen Wert oder eine Variable auf seinen Datentyp überprüfen. Zurückgegeben wird uns nur TRUE oder FALSE. Die für uns interessanten Funktionen hierfür heißen is.numeric(), is.character(), is.logical(), is.factor(), is.Date(), is.POSIXct() und is.na(). Natürlich gibt es auch is.double() und is.integer(), allerdings genügt uns für die Anwendungen in diesem Buch is.numeric(). Möchte man generell herausfinden, mit welchem Datentyp man es zu tun hat, verwendet man typeof().
[1] "character"Da es unpraktisch ist, bei diversen Spalten eines Datensatzes einzeln den Typ abzufragen, wird dieser in tibbles (siehe Kapitel 11.3) – dem Datensatzformat, welches wir innerhalb von R konsistent im gesamten Buch verwenden – direkt unter dem Spaltennamen angezeigt.
# A tibble: 200 × 7
Alter Geschlecht Extraversion Neurotizismus O1 O2 O3
<dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 36 m 3 1.9 5 1 5
2 30 f 3.1 3.4 5 3 5
3 23 m 3.4 2.4 3 3 5
4 54 m 3.3 4.2 2 5 3
# ℹ 196 more rowsDa dort nur die Spalten angezeigt werden, die auf den Bildschirm passen, bietet glimpse() eine übersichtlichere Möglichkeit, einen schnellen Überblick über sämtliche Datentypen zu erhalten.
Rows: 200
Columns: 7
$ Alter <dbl> 36, 30, 23, 54, 24, 14, 32, 20, 29, 17, 30, 15, 14, 23, 27, 15, 1964, 2…
$ Geschlecht <chr> "m", "f", "m", "m", "f", "f", "m", "m", "f", "m", "f", "f", "f", "m", "…
$ Extraversion <dbl> 3.0, 3.1, 3.4, 3.3, 3.0, 2.8, 3.5, 3.5, 3.0, 3.1, 3.2, 3.5, 3.0, 3.2, 2…
$ Neurotizismus <dbl> 1.9, 3.4, 2.4, 4.2, 2.8, 3.5, 3.1, 2.6, 3.7, 3.6, 3.6, 2.8, 3.8, 2.0, 3…
$ O1 <dbl> 5, 5, 3, 2, 5, 5, 3, 2, 4, 4, 5, 4, 2, 5, 4, 2, 5, 3, 5, 5, 3, 4, 2, 3,…
$ O2 <dbl> 1, 3, 3, 5, 1, 1, 1, 1, 1, 3, 2, 3, 3, 1, 2, 3, 1, 2, 4, 1, 1, 1, 4, 3,…
$ O3 <dbl> 5, 5, 5, 3, 5, 5, 5, 3, 5, 4, 5, 4, 3, 5, 4, 5, 5, 5, 4, 4, 1, 5, 3, 2,…Möchten wir nun mehrere Werte eines Datentypens aneinanderreihen, um sie zum Beispiel in eine Spalte eines Datensatzes zu schreiben, können wir c() (Abkürzung für Combine, engl. für kombinieren) verwenden. Möchtest du beispielsweise die Werte 1, 4, 5, und 10 kombinieren, erstellt dir c() einen entsprechenden Vektor (siehe Kapitel 11.1). Die Feinheiten und Merkmale von Vektoren brauchen dich an dieser Stelle nicht zu interessieren. Allerdings brauchen wir die Funktion c() des Öfteren, um Werte aneinander zu reihen.
Wenn du verschiedene Datentypen innerhalb von c() miteinander kombinierst, werden die Datentypen ineinander umgewandelt. Dabei gilt Character > Integer > Logical. Wenn Zahlen mit Buchstaben kombiniert werden, wird also alles zu Buchstaben, auch wenn eigentlich Zahlen gemeint sind. Es gibt diverse Datentypen, die auf diesen vier Grundtypen aufbauen. Zwei wichtige, Faktoren und Zeitdaten, werden wir uns im Folgenden noch anschauen.
Es kann passieren, dass Zahlen von R als Buchstaben interpretiert werden. Das kommt vor allem beim Einlesen von schlecht formatierten Datensätzen vor. Wenn eine Berechnung nicht so funktioniert, wie sie sollte, lohnt es sich, die Datentypen der jeweiligen Spalten zu überprüfen.
Übung 4.3.1. (TESTPHASE) Das Verstehen der Grunddatentypen ist zum Arbeiten mit R essentiell. In dieser Übung wird das Verständnis mithilfe allgemeiner Fragen und anhand einer Nutzerstatistik eines Musikstreamingdienstes geprüft. Besonders werden zudem die logischen Datentypen wiederholt. Starte die Übung mit uebung_starten("4-3-1") (siehe Kapitel 1.3).
4.3.2 Faktoren
Faktoren bieten in R die Möglichkeit, die Ausprägungsgrade von Kategorien eindeutig zu definieren und wahlweise auch in eine Reihenfolge zu bringen. Wir möchten Variablen (Spalten) also dann in einen Faktor umwandeln, wenn wir zwei oder mehr Kategorien vorliegen haben. Ein Faktor für zwei Kategorien erlaubt es uns beispielsweise die Reihenfolge der Balken in einem Balkendiagramm zu verändern. Bei mehr als zwei Kategorien können wir z.B. im Kontext von Regressionsmodellen sogenannte Dummy Variablen erstellen, die die Ausprägungen mit einer Referenzkategorie vergleichen.
Es handelt sich bei Faktoren also um nominalskalierte Kategorien, die in ausgewählten Anwendungsfällen auch geordnet werden können (ordinales Skalenniveau). Ein illustratives Beispiel hierfür wäre die Aufteilung in Experimental- und Kontrollgruppen. Bereits bei der Versuchsplanung überlegst du dir, wie viele Gruppen du benötigst (beispielsweise eine Experimental- und zwei Kontrollgruppen), um eine entsprechende Stichprobenplanung durchzuführen. Schauen wir uns das ganze nun etwas konkreter an. Angenommen, die Information über die Bedingung (Experimental, Kontrolle) befindet sich in der Variable namens Bedingung mit dem Datentyp Character.
Mit der Funktion factor() können nun Faktoren daraus gemacht werden. Zusätzlich sollte man das optionale levels Argument verwenden, um die Reihenfolge der Faktorstufen festzulegen.
[1] exp kont1 exp exp kont2 kont1
Levels: exp kont1 kont2Das levels Argument ist außerdem wichtig, wenn wir nicht alle Faktorstufen beobachtet haben. Wenn beispielsweise eine der Gruppen in der Erhebung nicht vorkam, möchten wir eine Häufigkeit von 0 angezeigt bekommen. Würden wir die Faktorstufen nicht explizit definieren, würde die fehlende Gruppe in den im weiteren Verlauf des Buches verwendeten Funktionen schlichtweg nicht angezeigt werden.
bedingung <- c("exp", "kont1", "exp", "exp", "kont1")
factor(bedingung, levels = c("exp", "kont1", "kont2"))[1] exp kont1 exp exp kont1
Levels: exp kont1 kont2Die Ausgabe der Funktion zeigt uns, dass die Stufe kont2 vorliegen könnte und innerhalb des Faktors gespeichert ist.
Wenn die als Faktor zu kodierende Spalte numerisch ist, können mit dem Argument labels die Namen der verschiedenen Ausprägungsgrade spezifiziert werden. Ein häufiges Beispiel hierfür wäre die Variable Geschlecht mit drei Ausprägungsgeraden.
Die Umkodierung geht mit dem labels Argument intuitiv, sofern man auf die Reihenfolge der Labels achtet.
[1] m m f d m d
Levels: m f dMithilfe des zusätzlich Arguments ordered ist es möglich, aus den nominalen Kategorien ein ordinales Skalenniveau zu bilden. Dies wäre z.B. bei der Untersuchung möglicher Zusammenhänge zwischen Schmerzintensität und verschiedenen Behandlungsmethoden notwendig (siehe Kapitel 9.5.4). Schließlich ist auf einer Schmerzskala von 0 (keine Schmerzen) bis 5 (unerträgliche Schmerzen) ein Schmerz von 3 als höher zu bewerten als 2. Da dennoch nicht bekannt ist, wie groß der Abstand zwischen den Stufen der Schmerzskala genau ist, haben wir hier ordinales Skalenniveau vorliegen (in Abgrenzung zu intervallskalierten Merkmalen). Angenommen, wir befragen fünf Personen über ihre momentan Schmerzen. Standardmäßig sind Faktoren nominal aufsteigend nach Größe sortiert.
[1] 0 3 1 5 4
Levels: 0 1 3 4 5Durch das ordered Argument erhalten diese eine explizite Reihenfolge.
[1] 0 3 1 5 4
Levels: 0 < 1 < 3 < 4 < 5Grundsätzlich sollte man Faktoren nur bei Bedarf erstellen, da Faktoren nicht mit allen Funktionen erwartungsgemäß harmonieren (z.B. unmittelbar vor der statistischen Analyse oder vor Erstellen einer Abbildung). Dies hängt damit zusammen, dass Faktoren als Zahlen gespeichert werden. So erhalten wir für den Faktor fact1 als class() den Datentyp des Faktors,
[1] "factor"während uns typeof() den Datentyp Integer zurückgibt.
[1] "integer"Wie man mit Faktoren konkret umgehen und diese verändern kann, wird in Kapitel 6.10 erklärt.
Tatsächlich behandelt R Faktoren als Integer (Zahlen), was zu überraschenden Outputs führen kann. Verwende Faktoren also am besten nur dann, wenn du sie wirklich brauchst. Beispielsweise zum Rechnen inferenzstatistischer Verfahren oder unmittelbar vor dem Erstellen von Visualisierungen.
Übung 4.3.2. (TESTPHASE) In dieser Übung werden die typischen Verständnisprobleme im Kontext von Faktoren näher beleuchtet und die Konzepte anhand eines Datensatzes zur statistischen Bildung wiederholt. Starte die Übung mit uebung_starten("4-3-2") (siehe Kapitel 1.3).
4.3.3 Zeitdaten
Wie bereits in Abbildung 4.6 illustriert, interessieren uns die Datentypen Date und POSIXct. Letzteres ist im Regelfall ein unerwünschtes Format, welches häufig beim Einlesen von Excel Dokumenten entsteht.
Der Datentyp POSIXct ist ein Akronym für Portable Operating System Inferface calendar time (in der Ausgabe von tibbles und der Funktion glimpse() mit dttm für Datetime abgekürzt). Enthalten ist das Datum (Jahr.Monat.Tag) und die Uhrzeit. Die Uhrzeit ist allerdings in den wenigstens Forschungskontexten von Interesse.
[1] "2024-01-09 08:30:00 CET"In das gewünschte Date Format können wir mit as.Date() umwandeln.
[1] "2024-01-09"Hier haben wir nur Informationen über das Jahr, den Monat und den Tag. Wenn wir ein Datum ausgeben, sieht es zunächst wie eine Buchstabenfolge aus.
[1] "2002-02-15"Wir können uns aber mit is.Date() vergewissern, dass wir den Datentype Date vorliegen haben.
[1] TRUEÄhnlich wie bei Faktoren müssen wir auch hier zum Verständnis zwischen der Klasse des Datums
[1] "Date"und dem Datentyp unterscheiden.
[1] "double"Der zugrundeliegende Datentyp des Datums ist eine Dezimalzahl (Double). Intern speichert R das Datum als Anzahl von Tagen seit einem bestimmten Datum ab. Meistens ist dieses Datum der 1. Januar 1900. Von diesem Datum bis zum 15. Februar 2002 sind 37300 Tage vergangen. Überprüfen können wir das durch Subtrahieren der beiden Daten.
Time difference of 37300 daysDieses Format ist eine sogenannte difftime (engl. für Zeitdifferenz). Es passiert tatsächlich öfter als man denkt, dass das Datum nicht als Datum sondern als Zahl angezeigt wird. Dann können wir unter Angabe des Startdatums (origin), die Zahl wieder in ein Datum umwandeln.
[1] "2002-02-15"Die Gründe hierfür können vielfältig sein. Wenn man bspw. bei unbekanntem Tag des Datums "UN.06.2022" in eine Zelle einträgt. Beim Einlesen kann die Spalte dann nicht als Datum eingelesen werden. Dies zu korrigieren, kann unter Umständen sehr mühsam sein. Einige Methoden zum konkreten Umgang mit diesen und weiteren Problemstellungen im Kontext von Zeitdaten wird in Kapitel 6.11 erläutert.
Übung 4.3.3. (TESTPHASE) An dieser Stelle kannst du dein Verständnis der hier kennengelernten Zeitdatentypen anhand mehrerer Fragen wiederholen. Mit einem Datensatz zur Nutzerstatistik einer Videostreaming Plattform wird das Gelernte weiter konkretisiert. Starte die Übung mit uebung_starten("4-3-3") (siehe Kapitel 1.3).
4.3.4 Datentypen konvertieren
Datentypen können auch umgewandelt werden. Die Namen sind ähnlich wie beim Abfragen des Datentypes, nur dass der Präfix hier nicht is, sondern as ist. Um den vorhin erstellten Vektor vec in den Typ Character umzuwandeln, würde man dementsprechend as.character() verwenden.
`
[1] "1" "4" "5" "10"Wie man sieht, stehen nun sämtliche Zahlen in Anführungszeichen, weswegen man zum Beispiel nicht mehr eine Zahl ohne Weiteres addieren könnte.
as.numeric()as.character()as.factor()as.Date()
Beachte hier, dass nur bei Date ein Großbuchstabe verwendet wird. Wenn leere Character in Numerics umgewandelt werden, generiert R automatisch fehlende Werte (NAs).
4.4 Struktur von Datensätzen
Bis auf wenige Ausnahmen setzen sämtliche im Buch verwendete Funktionen eine bestimmte Struktur der Daten voraus:
- Jede Zeile bezieht sich auf eine Beobachtung (z.B. jede Zeile enthält die Werte von einem Proband oder einer Patientin).
- Jede Spalte enthält eine Variable (z.B. Alter, Familienstand oder Überlebenszeit).
- Jede Zelle beinhaltet einen einzigen Wert, der genau einer Variable einer Beobachtung zuzuordnen ist.
Wenn diese Voraussetzungen erfüllt sind, spricht man auch von einem tidy (engl. für aufgeräumten) Datensatz. Achte also am besten bereits beim Erheben der Daten auf eine richtige Struktur, da sonst die Funktionen, die wir in Kapitel 6 kennenlernen werden, nicht ohne Weiteres anwendbar sind. Als ein Praxisbeispiel betrachten wir den big5 Datensatz. Darin entspricht jede Zeile einem Proband oder einer Probandin, der oder die einen Fragebogen zu den Big5 Persönlichkeitsfaktoren beantwortet haben. Die Spalten sind dabei eine Mischung aus demographischen Variablen (z.B. Alter) und den Werten der eigentlichen Fragen (z.B. O1 für die erste Frage der Dimension Offenheit für neue Erfahrungen).
# A tibble: 200 × 7
Alter Geschlecht Extraversion Neurotizismus O1 O2 O3
<dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 36 m 3 1.9 5 1 5
2 30 f 3.1 3.4 5 3 5
3 23 m 3.4 2.4 3 3 5
4 54 m 3.3 4.2 2 5 3
# ℹ 196 more rows4.5 Der Dollar-Operator
Manche Funktionen benötigen jedoch nicht den gesamten Datensatz im vorherigen Kapitel beschriebenen Format als Argument, sondern lediglich eine Spalte daraus. Diese Spalte können wir mit dem sogenannten Dollar-Operator aus dem Datensatz extrahieren. Das Praktische an dem Dollar-Operator im Vergleich zu anderen Methoden zum Extrahieren der Spalte ist die Möglichkeit der automatischen Vervollständigung durch RStudio.
Wenn wir den Namen des Datensatzes big5 gefolgt von einem Dollar-Zeichen angeben, werden uns alle enthaltenen Spalten in einem Dropdown-Menu angezeigt. Dies ist vor allem bei langen Namen der jeweiligen Spalten äußerst praktisch und schützt uns vor Tippfehlern. Hier wählen wir exemplarisch die Spalte Alter aus.
Dieser Befehl würde uns alle 200 Alterswerte in einer Wertereihe bzw. als sogenannter Vektor zurückgegeben werden (siehe Kapitel 11.1). Um das ganze übersichtlich zu halten, lassen wir uns mit head() nur das Alter der ersten 6 Personen ausgeben.
[1] 36 30 23 54 24 14Als zweites Beispiel schauen wir uns den demogr Datensatz an, in welchem die drei Spalten ID, Sex und Alter enthalten sind.
# A tibble: 4 × 3
ID Sex Alter
<chr> <chr> <dbl>
1 AX161095 m 28
2 NM020683 f 47
3 IO240576 f 40
4 JH051199 m 24Die einzelnen Angaben über das biologische Geschlecht der Personen innerhalb des Datensatzes, werden erneut mit der Kombination aus Datensatznamen, Dollar-Zeichen und Spaltennamen als Aneinanderreihung von Werten extrahiert.
[1] "m" "f" "f" "m"Nach demselben Prinzip können wir auch die Personenkennungen aus der Spalte ID ausgeben lassen. Auch hier werden nur vier Werte zurückgegeben, da im demogr Datensatz die Informationen von vier Personen gespeichert sind.
[1] "AX161095" "NM020683" "IO240576" "JH051199"