Kapitel 5 Datensätze
Mit einer Funktion alle erdenklichen Dateiformate einzulesen, klingt fast zu schön, um wahr zu sein. Durch ein Package ist dies in R tatsächlich bereits lange Realität. Neben den Einlesen externer Datensätze wird außerdem gezeigt, wie man Datensätze aus Packages laden oder direkt in R erstellen kann. Auch das Abspeichern der in R modifizierten Daten wird abschließend erklärt.
5.1 Einlesen externer Dateien
Weißt du, was Projekte sind und kannst diese innerhalb von RStudio erstellen? Wenn nicht, lies dir das Konzept der Projekte in R genau durch (siehe Kapitel 3.3). Ansonsten kann R die Datei mit deinem Datensatz nicht finden und somit auch nicht einlesen. Es muss also immer zunächst ein neues R Projekt erstellt werden. Der Datensatz muss sich dabei im gleichen Ordner wie die Projektdatei befinden.
Im Regelfall möchte man den Datensatz nicht direkt in R erstellen, sondern einen bereits existierenden Datensatz zur Auswertung einlesen. Datensätze können dabei in verschiedenen Formaten vorliegen. Dies ist vor allem abhängig davon, mit welchen Programmen Unternehmen, Universitäten oder KollegInnen zur Datenerhebung arbeiten. Einige Beispiele für Dateientypen, in denen Daten häufig gespeichert werden, sind:
- R (
.RData|.rda|.rds) - Excel (
.xlsx|.xls) - SPSS (
.sav) - Stata (
.dta) - Tabular separated values (
.tsv) - Comma separated values (
.csv) mit Punkt als Dezimaltrenner oder mit Komma (siehe Infobox)
Sämtliche Datentypen können wir mit import() aus dem rio Package einlesen. Wir laden also zunächst das Package.
Um auch exotischere Dateiformate einlesen zu können, solltest du einmal den Befehl install_formats() ausführen. Es ist generell empfohlen, diese Funktion einmalig auszuführen, da sonst jedes Mal beim Laden des Packages eine entsprechende Meldung angezeigt wird.
Wir möchten an dieser Stelle einen Datensatz über die Big 5 Persönlichkeitsfaktoren einlesen, welcher in Form eines Excel Dokument auf dem Computer gespeichert ist (siehe Abbildung 5.1).
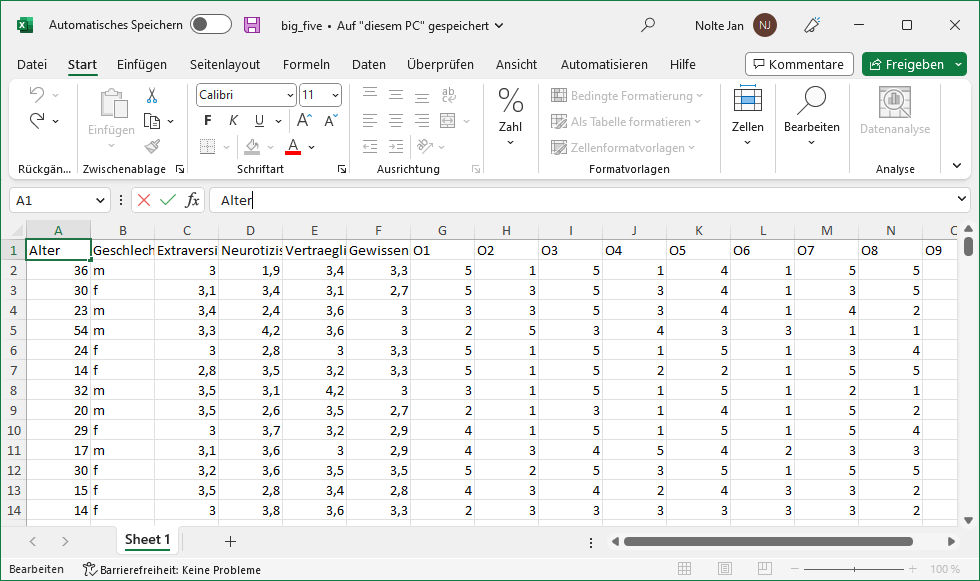
Abbildung 5.1: Big Five Datensatz als Excel Dokument.
Beim Einlesen erkennt die Funktion import() die Art der Datei anhand der Endung und übernimmt hinter den Kulissen alles Weitere. Damit der Datensatz als sogenannter tibble eingelesen wird, solltest du zusätzlich das Argument setclass = "tbl" setzen. Weshalb wir die Sonderform der tibbles verwenden und was dies genau ist, wird im Verlaufe des Buches klar. Für den Moment musst du dir beim setclass Argument allerdings noch nichts denken, es aber trotzdem verwenden.
Würden wir nur die import() Funktion aufrufen, würde der Datensatz zwar eingelesen, aber sofort wieder verschwinden, da dieser in R nicht abgespeichert wäre. Daher müssen wir den eingelesenen Datensatz, so wie in Kapitel Kapitel 4.2 beschrieben, einer Variable zuordnen. Der Name dieser Variable ist dabei nicht wichtig. Falls mit mehr als einem Datensatz gearbeitet wird, sollte ein aussagekräftigerer Name als der hier verwendete Name daten verwendet werden.
Der Name des Datensatzes muss dabei in Anführungszeichen gesetzt werden. In unserem Beispiel heißt das Excel Dokument big_five.xlsx. Damit wir den Datensatz in R angezeigt bekommen, müssen wir den Namen des gespeicherten Datensatzes anschließend erneut ausführen (siehe Kapitel 4.2). Dabei wird der Variablenname (hier daten) und nicht der Name der eigentlichen Datei (hier big_five.xlsx) verwendet.
# A tibble: 200 × 16
Alter Geschlecht Extraversion Neurotizismus Vertraeglichkeit Gewissenhaftigkeit O1 O2
<dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 36 m 3 1.9 3.4 3.3 5 1
2 30 f 3.1 3.4 3.1 2.7 5 3
3 23 m 3.4 2.4 3.6 3 3 3
4 54 m 3.3 4.2 3.6 3 2 5
# ℹ 196 more rows
# ℹ 8 more variables: O3 <dbl>, O4 <dbl>, O5 <dbl>, O6 <dbl>, O7 <dbl>, O8 <dbl>, O9 <dbl>,
# O10 <dbl>Alternativ könnte man zur expliziten Ausgabe beim Speichern auch Klammern um den ganzen Befehl setzen.
Die Datei (hier das Excel Dokument) muss sich innerhalb desselben Ordners wie die Projektdatei befinden. Falls die Datei in einem Unterordner ist, muss man den relativen Pfad – also den Weg bis zur Datei innerhalb der Unterordner (hier namens Daten) des Ordners (hier namens Beispiel) – zusätzlich der R Funktion mitteilen.
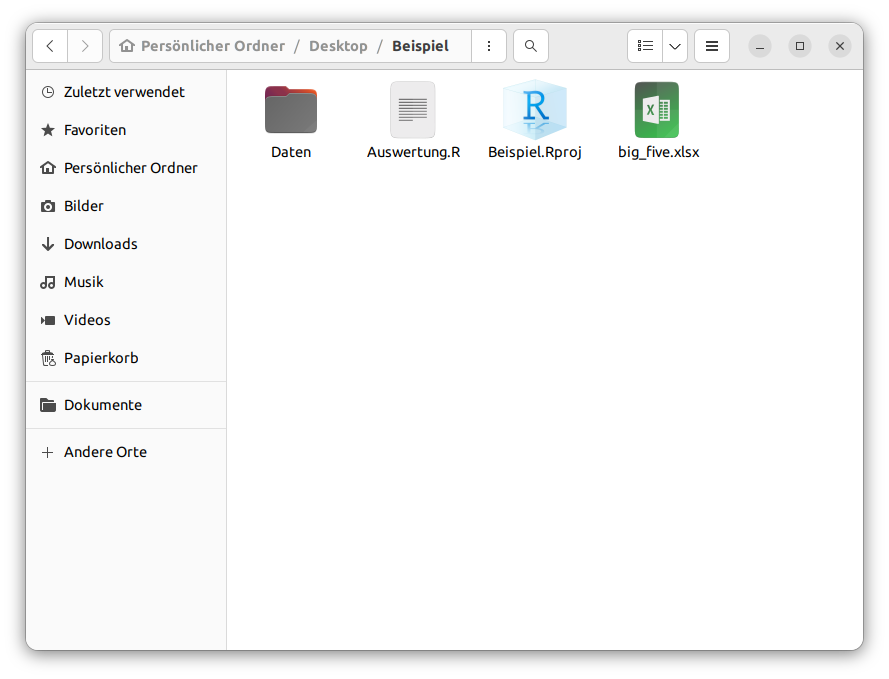
Abbildung 5.2: Beispielhafte Ordnerstruktur mit Unterordnern für Datensätze, R Skript und Projektdatei.
Dabei nutzen wir die Funktion here() aus dem gleichnamigen Package. Dort können wir als separate Argumente den gesamten relativen Pfad eintragen. In diesem Beispiel liegt innerhalb des Ordners Daten der Datensatz bildung.xlsx.
Ein weiteres nützliches Argument stellt skip dar, mit dem Zeilen beim Einlesen ignoriert werden. Besonders in Excel Dokumenten werden häufig zusätzliche Überschriften für die Variablennamen verwendet. Das mag zwar in Excel übersichtlich aussehen, allerdings weicht dies von der in Kapitel 4.4 kennengelernten Datenstruktur ab und macht deshalb beim Einlesen Probleme.
Ein Sonderfall liegt beim Einlesen von R Datensätzen mit den Endungen .rds oder .RData vor, da wir hier zusätzlich das Argument trust auf TRUE setzen müssen. Das hat den Hintergrund, dass bei fremden Datensätzen in diesem Format Schadsoftware auf den Computer gelangen kann.
Manchmal liegen Daten nicht in einer, sondern in vielen verschiedenen Dateien vor. Das ist vor allem häufig bei biophysiologischen Messungen wie beim Eye Tracking der Fall, bei denen die erhobenen Daten pro Person abgespeichert werden. Da wir nicht 20 Mal import() kopieren möchten (da Copy & Paste sehr fehleranfällig ist), gibt es die Funktion import_list(). Angenommen, im Ordner Daten wären unsere 20 Excel Dokumente, in denen jeweils die Daten pro Person liegen. Dann könnte man zuerst mit dir() die Dateinamen herausfinden, um diese dann mit import_list() einzulesen. Mit dem Zusatzargument rbind = TRUE können wir die Datensätze direkt zusammenfügen. Voraussetzung dafür ist natürlich, dass die Datensätze dieselben Spalten haben. Da die Dateien in einem Unterordner liegen, müssen wir import_list() zusätzlich mit der Funktion here() mitteilen, wo sich die Datensätze befinden.
dateien <- dir("Daten", pattern = ".xlsx$")
daten <- import_list(
file = here("Daten", dateien),
setclass = "tbl",
rbind = TRUE
)Das Dollar-Zeichen signalisiert in diesem Fall nur, dass wir am Ende des Dateinamens die Endung .xlsx (also ein Excel Dokument) erwarten. Dasselbe funktioniert übrigens für den Fall, wenn sich in Excel mehrere Arbeitsblätter innerhalb eines Dokuments befinden. Mit import_list() können alle auf einen Schlag eingelesen werden.
Die verschiedenen Datensätze sind dann in einer Liste gespeichert (siehe Kapitel 11.4). Hätten wir in einer Arbeitsmappe die Blätter Demographie und Messungen könnten wir nach Einlesen mit import_list() mithilfe des Dollar Operators in Form von daten$Demographie und daten$Messungen auf die Datensätze in der Liste daten zugreifen.
Alternativ kann man im Fall von Excel Arbeitsmappen mit mehreren Arbeitsblättern mit dem Argument which in der Funktion import() auswählen, welches Arbeitsblatt eingelesen werden soll. In diesem Fall erhalten wir einen normalen Datensatz ohne Listenstruktur. So könnte man die zweite Arbeitsmappe namens Messungen aus dem obigen Beispiel mit import("Datensaetze.xlsx", setclass = "tbl", which = 2) aus der Arbeitsmappe namens Datensaetze einlesen.
Die in diesem Kapitel kennengelernten Funktionen können wir praktisch für alle Dateienarten verwendet werden. Falls also anders als in unserem Beispiel die Daten nicht in einem Excel Dokument, sondern einer Datei mit der Endung .csv enthalten sind, müssen wir lediglich die Endung verändern (z.B. big_five.csv).
Im deutschsprachigen Raum werden Dezimalzahlen meistens durch ein Komma anstelle eines Punktes getrennt. Dies ist vor allem im Kontext von CSV Dateien problematisch, da hier in allen anderen Teilen der Welt das Komma zur Trennung der einzelnen Spalten bzw. Variablen verwendet wird. Um trotzdem ein fehlerfreies Einlesen gewährleisten zu können, benötigen wir innerhalb der Funktion import() das zusätzliche Argument format = "csv2". Der vollständige Befehl könnte also wie folgt aussehen: import("big_five.csv", setclass = "tbl", format = "csv2").
5.2 Datensätze aus Packages laden
Zum Bearbeiten dieses Buches brauchst du keine externen Datensätze, sondern nur jene, die im remp Package enthalten sind. So kannst du das Gelernte sofort begleitend in RStudio nachvollziehen und ausprobieren. Nach Laden des Packages hast du grundsätzlich sofort Zugriff auf alle enthaltenden Datensätze, allerdings werden diese erst nach Aufrufen der Funktion data() in R gespeichert. Zum Einlesen des big5 Datensatzes, müsste man demnach lediglich den Namen des Datensatzes der Funktion data() übergeben.
Sollte das Package vorher nicht geladen sein, kann alternativ auch zusätzlich das Argument package zur Spezifizierung des Ursprungs des Datensatzes verwendet werden.
Da der Befehl etwas länger ist und beim Üben das remp Package sowieso geladen werden sollte, ist allerdings der zuvor dargestellte Weg empfohlen.
5.3 Datensätze in R erstellen
Direkt innerhalb von R Datensätze zu erstellen, ergibt nur in wenigen Anwendungsfällen wirklich Sinn. Der womöglich Wichtigste ist das Erstellen eines minimalen reproduzierbaren Beispiels, falls man auf einen Fehler stößt, den man selbst nicht lösen kann. Für größere Datensätze sollte man die Daten jedoch besser in Datenformaten wie .csv oder .xlsx kreieren.
Möchte man einen Datensatz erstellen, muss man lediglich der Funktion tibble() Werte übergeben. In diesem Beispiel speichern wir den neuen Datensatz mit den zwei Spalten Extraversion und Geschlecht als my_tbl. Die Funktion c() (engl. für combine) kombiniert die einzelnen Werte eines Datentyps und kettet selbige aneinander.
# A tibble: 4 × 2
Extraversion Geschlecht
<dbl> <chr>
1 1.2 m
2 2.7 f
3 1.5 f
4 4.8 m 5.4 Speichern und Konvertieren
Das Speichern von Datensätzen funktioniert durch das rio Package ähnlich intuitiv wie das Importieren von Dateien. Anstelle von import() benutzen wir dafür stattdessen export(). Das erste Argument der Funktion ist der Datensatzname und das zweite Argument ist der gewünschte Dateiname. Der Dateientyp wird durch die gewählte Endung festgelegt. Möchte man beispielsweise den fertig aufbereiteten Datensatz video_clean als csv Datei abspeichern, würde man
schreiben. Manchmal ist es nützlich, den Datensatz unabhängig vom Einlesen umzuwandeln. Das kann zum Beispiel der Fall sein, wenn deine Kollegen dir eine SPSS Datei schicken (.sav) und du selbst kein SPSS hast, aber trotzdem einen Blick in die Daten werfen möchtest. Die Entwickler des rio Packages haben auch daran gedacht und die Funktion convert() geschrieben. Als erstes Argument übergibst du der Funktion den ursprünglichen Dateinamen (mit Endung) und als zweites denselben oder einen anderen Dateinamen mit der Endung des gewünschten Dateientypen. Sinnvoll wäre in diesem Kontext das Umwandeln in ein Excel Dokument, da dieses mit MS oder Libre Office problemlos geöffnet werden kann.
Die neue Datei wird sowohl bei export() als auch bei convert() in deinem Projektverzeichnis gespeichert.
Besonders wichtig ist auch das Abspeichern von kategorealen Variablen als Buchstaben oder Wörter (Datentyp Character) und nicht als Zahlen. Nach spätestens einem Jahr kann kein Mensch mehr nachvollziehen, ob beispielsweise eine 1 nun für weiblich und 0 für männlich oder eine 1 für männlich und eine 0 für weiblich verwendet wurde.
# A tibble: 4 × 2
Extraversion Geschlecht
<dbl> <dbl>
1 1.2 0
2 2.7 1
3 1.5 1
4 4.8 0Stattdessen sollte man die Zahlen in Character umwandeln. Wie das geht, wird in Kapitel 6.4.3 gezeigt.
# A tibble: 4 × 2
Extraversion Geschlecht
<dbl> <chr>
1 1.2 m
2 2.7 f
3 1.5 f
4 4.8 m Stelle vor dem Abspeichern des Datensatzes immer sicher, dass alle Variablen unmissverständlich kodiert sind. Kategorien sollten immer explizit benannt werden.